基于舌苔微生物的肿瘤预测系统的应用的制作方法-j9九游会真人
基于舌苔微生物的肿瘤预测系统的应用
1.本技术是申请日“2022.07.22”、申请号“202210868365.0”、发明名称“基于舌苔微生物的肿瘤预测系统、方法及其应用”的中国发明专利申请的分案申请。
技术领域
2.本发明涉及肿瘤学诊断、预测、评估技术领域,更具体来说,涉及基于舌苔微生物的肿瘤预测系统、方法及其应用,通过分析舌苔微生物与肿瘤学的关联关系,从而实现经济的、非侵入性的且具有较高准确度的肿瘤预测。
背景技术:
3.根据最新数据,胃癌(gc)是全球第三大癌症相关死亡原因。gc的诊断和筛查仍然依赖于胃镜检查,但由于其侵入性强、成本高以及需要专业的内镜医师,其应用受到很大限制。此外,由于胃癌早期缺乏特异性症状,临床疾病标志物的特异性和敏感性较差,超过60%的患者在确诊时即发生局部或远处转移。局部早期gc患者的5年生存率超过60%,而局部、远处转移患者的5年生存率分别显著下降至30%和5%。因此,迫切需要新的gc诊断或筛查方法,以提高该人群的早期诊断率和预后效果。
4.中医药是几千年来中国人民经验应用和保留的医学科学和文化遗产,舌象诊断是中医诊断疾病的重要依据之一。中医理论认为,舌象的变化(舌的颜色、大小和形状,舌苔的颜色、厚度和含水量)可以反映人体的健康状况,尤其与胃病密切相关。在中医胃病诊断中,往往根据舌象信息采取经验或辩证的方式得出胃病的具体表现形式,又有研究表明口腔或者舌苔微生物群密切相关。
5.人工智能(ai)可用于筛查、诊断和治疗各种疾病,cheung cy等学者的论文(cheung cy,xu d,cheng cy,et al.a deep-learning system for the assessment of cardiovascular disease risk via the measurement of retinal-vessel calibre.nature biomedical engineering 2021;5(6):498-508.doi:10.1038/s41551-020-00626-4[published online first:2020/10/14])公开了一种深度学习系统,通过测量视网膜血管的口径来评估心血管疾病的风险,可以有效预测心血管疾病的风险。takenaka k等学者的论文(takenaka k,ohtsuka k,fujii t,et al.development and validation of a deep neural network for accurate evaluation of endoscopic images from patients with ulcerative colitis.gastroenterology 2020;158(8):2150-57.doi:10.1053/j.gastro.2020.02.012[published online first:2020/02/16])公开了一种深度神经网络(见参考文献),用于评估溃疡性结肠炎患者的内窥镜图像,该网络以90.1%的准确度识别内镜缓解和组织学缓解的患者,准确率为92.9%。
[0006]
福州数据技术研究院有限公司专利cn110251084a提供一种基于人工智能的舌像检测与识别方法,用于解决舌像采集过程中舌像舌体的实时检测拍摄、保存、上传,同时识别舌像舌色、舌形、苔质、苔色;其方案主要涉及了舌像的采集和识别技术,其中舌像识别更侧重于提取舌像颜色、纹理、舌苔区域或舌苔厚薄等特性,然而这些工作并没有将舌像、舌
苔信息与某一特殊胃病比如胃癌建立对应关系。
[0007]
沈阳智朗科技有限公司专利cn111710394a提出一种人工智能辅助的早期胃癌筛查系统,以自动化代替人工分析胃镜切片图像来解决胃癌阳性确定工作量大的问题;然而此种基于胃镜图像分析的策略,仍然首先需要获得大量的专业仪器采集的胃镜图像用于模型的学习,在测试阶段仍然需要依据每个测试者的胃镜图像做出决策,而胃镜图像的获得仍然存在时间消耗大、物质成本高、测试人群标准高等缺陷,很难做到全国范围的普查筛选。
[0008]
江苏天瑞精准医疗科技有限公司专利cn112133427a提供了一种基于人工智能的胃癌辅助诊断系统,包括:诊断选择模块、数据采集模块、预处理模块、诊断模块和显示输出模块,该系统能够根据采集到的就诊者的数据,个性化地给出诊断结果。该诊断系统诊断所依据的数据包括就诊者的基本信息、生活饮食、感染史、疾病史、家族史、临床症状和检验项目等,其中临床症状和检验项目等数据的收集难度较大,而单独依靠基本信息、生活饮食、感染史、疾病史、家族史等信息则会影响前期的筛查诊断效果。
[0009]
上海仁东医学检验所有限公司专利cn114203256a提供了一种基于微生物丰度的mibc分型及预后预测模型构建方法,该方法主要是从tcga数据库中的mibc转录组rna-seq数据中分析得到mibc(肌层浸润性膀胱癌)患者的微生物数据,然后以微生物丰度谱为特征进行nmf聚类,建立mibc微生物层面的分子分型,从而从肿瘤组织微生物层面深度解析了微生物与mibc的相关性,建立了mibc预后预测模型,该模型有助于精准预测mibc患者的1-5年生存率。因此该发明方案主要是为在微生物层面为mibc建立分子分型,目的是精准预测患者预后生存率。
[0010]
然而遗憾的是目前尚未有研究证实舌苔微生物种群的变化与肿瘤存在对应关系,以及舌苔微生物种群变化在肿瘤诊断和筛查中的价值。
[0011]
本发明力图解决这些和本领域中的其他待解决的需要。
技术实现要素:
[0012]
为解决上述背景技术中提及的至少一种技术问题,本发明旨在利用计算机辅助手段,设计一种基于深度学习的肿瘤筛查系统,该系统根据舌苔微生物中不同种、属丰度自动化预测不同测试试样属于肿瘤阳性的概率,以此来作为一种经济的、非侵入性的、高效的、准确的肿瘤早期筛查策略。
[0013]
基于舌苔微生物的肿瘤预测系统,包括:
[0014]
微生物信息获取模块,其被配置为获取测试试样的舌苔微生物信息;
[0015]
数据处理模块,其被配置为通过下述操作来获得测试试样属于肿瘤阳性的概率:
[0016]
依据自动学习获得的舌苔微生物信息上的可判别性的特征预测测试试样属于阳性的概率。
[0017]
在一个具体实施例中,所述肿瘤是胃癌、乳腺癌、结直肠癌、食道癌、肝胆胰腺癌、肺癌、前列腺癌、甲状腺癌、卵巢癌、神经母细胞瘤、滋养细胞肿瘤或头颈部鳞癌中的至少一种。
[0018]
在一个具体实施例中,所述肿瘤是胃癌、乳腺癌、结直肠癌、食道癌、肝胆胰腺癌或肺癌中的至少一种。
[0019]
在一个具体实施例中,所述肿瘤是胃癌。
[0020]
在一个具体实施例中,所述系统还包括输出模块,其被配置为输出预测结果。
[0021]
在一个具体实施例中,所述输出模块被配置为输出舌苔微生物信息与预测结果。
[0022]
在一个具体实施例中,所述输出模块以电子显示、声音播报、打印、网络传输的至少一种模式输出。
[0023]
在一个具体实施例中,所述舌苔微生物信息包括舌苔微生物的属、种丰度。
[0024]
在一个具体实施例中,所述可判别性的特征来自于微生物的属、种丰度。
[0025]
在一个具体实施例中,所述可判别性的特征来自于微生物的属、种丰度的高维特征。旨在通过充分对比、分析、学习阳性患者与阴性患者之间关于舌苔微生物信息的共性和差异,通过深度判别测试试样舌苔微生物属、种丰度之间可判别性的特征,获得阳性患者和阴性患者之间的差异,即可判断得出测试试样属于肿瘤阳性的概率,从而通过对舌苔微生物信息的对比、分析、学习即可实现对测试试样肿瘤的诊断和预测,提供一种非侵入性的、非以人体组织为来源的、经济性高、准确率高的肿瘤诊断预测系统。
[0026]
在一个具体实施例中,所述数据处理模块通过下述操作来获得测试试样属于肿瘤阳性的概率:
[0027]
训练完成的神经网络对输入其中的微生物属、种丰度提取高维特征后预测测试试样属于阳性的概率。
[0028]
在一个具体实施例中,所述神经网络是多层感知器(mlp)。
[0029]
在一个具体实施例中,所述神经网络以下述步骤训练:
[0030]
1)将采集自肿瘤阳性患者和/或肿瘤阴性人群的舌苔微生物属和/或种丰度作为输入向量输入至模型的输入层;
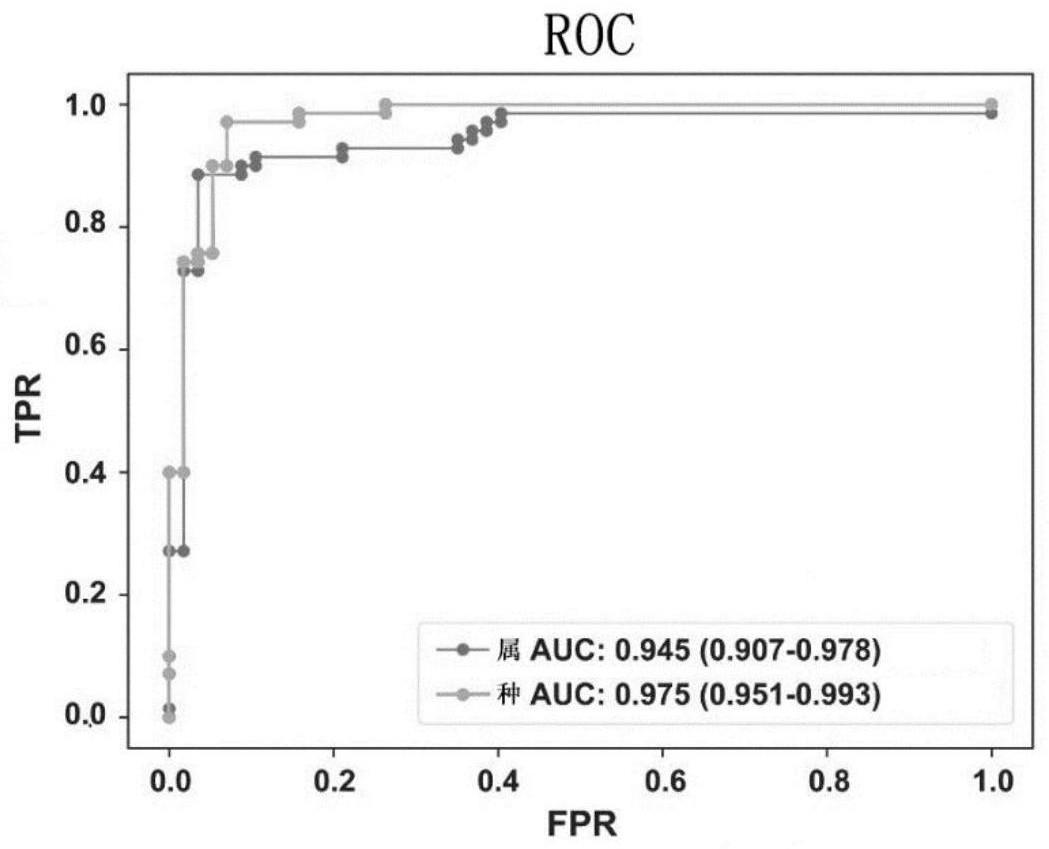
[0031]
2)模型的隐藏层提取微生物种和/或属丰度的高维特征;
[0032]
3)通过输出层的softmax分类器输出舌苔微生物属和/或种分属阳性和阴性的概率分布。
[0033]
在一个具体实施例中,所述输入向量的长度是706/1339,分别代表微生物属/种。
[0034]
在一个具体实施例中,所述输入向量的每一个元素代表在特定属或种上的微生物丰度。
[0035]
在一个具体实施例中,所述输入层和输入向量长度对应。
[0036]
在一个具体实施例中,所述隐藏层的神经元个数设置为512。隐藏层神经元个数大于输入层神经元个数,所以在前向运算时,将输入层的丰度特征非线性映射到高维空间中,形成高维特征,提取获得高维特征后即可精油输入层输出分属阳性和阴性的概率。
[0037]
在一个具体实施例中,所述神经网络中除了输出层之外的每一层都附带激活函数和归一化。
[0038]
在一个具体实施例中,所述输出层包括两个神经元:肿瘤阳性和肿瘤阴性。
[0039]
在一个具体实施例中,所述神经网络的各层神经元分别互相连接,利用输出的概率分布最小化交叉熵目标函数:
[0040][0041]
其中yi是测试样例对应的真实one-hot标注向量中的元素,是模型预测为类别yi的概率,其中0代表阴性类别,1代表阳性类别。one-hot标注是0、1向量形式的标注,比如分两个类别,类别0,1所对应的one-hot形式的标注是(1,0,0),(0,1,0)。
[0042]
经过大量真实患者的验证,表明应用舌苔微生物作为无创诊断和筛查肿瘤的手段明显优于常规的血液肿瘤标志物,基于舌苔微生物的肿瘤预测系统具有比常规基于血液肿瘤标志物的人工智能模型更为优异的属/种敏感性(0.914/0.929vs 0.283-0.566、0.362-0.539)、特异性(0.947/0.947vs 0.688-0.976、0.759-0.938)和准确性(0.929/0.937vs 0.603-0.622、0.645-0.662),其auc值也更高(0.945/0.975vs 0.682-0.715、0.694-0.760),考虑到中国及全球肿瘤检测的巨大负担,认为舌苔微生物结合人工智能深度学习方法的广泛使用,是筛查和预测早起肿瘤的最经济的、非侵入性的和可接受的方法,这也将带来巨大的社会经济影响。
[0043]
增加隐藏层数量或者调整隐藏层单元个数也可以达到相近的识别精度,即由不同超参数的全连接层组成的神经网络都适用于基于微生物丰度的肿瘤阳性判别任务,并通过深度学习技术,自动化判断肿瘤阳性的概率,以筛选出肿瘤高发人群。
[0044]
基于舌苔微生物的肿瘤预测方法,其包括:
[0045]
获得测试试样的舌苔微生物信息;
[0046]
将测试试样的舌苔微生物信息输入前述所述系统获得所述测试试样的肿瘤阳性概率。
[0047]
所述舌苔微生物信息包括舌苔微生物的属、种丰度。
[0048]
前述所述基于舌苔微生物的肿瘤预测系统和/或方法的应用,其包括:
[0049]
应用所述系统和/或方法对测试试样进行肿瘤预测。
[0050]
在符合本领域常识的基础上,上述各优选条件,可以相互组合,得到具体实施方式。
[0051]
本发明的有益效果为:
[0052]
本发明旨在提供一种基于深度学习的肿瘤筛查系统,通过充分对比、分析、学习阳性患者与阴性患者之间关于舌苔微生物信息的共性和差异,获得阳性患者和阴性患者之间的差异,通过深度判别测试试样舌苔微生物属、种丰度之间可判别性的特征即可判断得出测试试样属于肿瘤阳性的概率,从而通过对舌苔微生物信息分析学习即可实现对测试试样肿瘤的诊断和预测,验证得知舌苔微生物预测胃癌时的属/种敏感性达0.914/0.929,特异性均达0.947,准确性达0.929/0.937,其auc值达0.945/0.975,预测效果比常规基于血液肿瘤标志物的人工智能模型更加优异,可以此来作为一种经济的、非侵入性的、高效的、准确的肿瘤早期筛查策略,必将带来巨大的社会经济效益。
[0053]
本发明为实现上述目的而采用了上述技术方案,弥补了现有技术的不足,设计合理,操作方便。
附图说明
[0054]
旨在为使得本领域技术人员更加迅速明确的了解本技术的上述和/或其他目的、特征、优点与实例,提供了部分附图,应当指出的是,构成本技术的说明书附图、示意性实施例及其说明用来提供对本技术的进一步理解,并不构成对本技术的不当限定。
[0055]
图1是多中心临床研究及其病患分布示意图;
[0056]
图2是本技术神经网络的整体算法结构;
[0057]
图3是基于8个血液肿瘤的三种模型(svm、dt、knn)内部验证及外部验证的roc和auc;
[0058]
图4是gcs组和ngcs组的微生物主坐标分析图;
[0059]
图5是gcs组和ngcs组前30个微生物的热图;
[0060]
图6是香农多样性指数;
[0061]
图7是基于舌苔微生物的肿瘤预测系统对验证集的roc和auc;
[0062]
图8是gcs组和ngcs组的微生物物种分布图(其中e是微生物物种分布在属水平上的显示,f是微生物物种分布在种属性上的显示)。
具体实施方式
[0063]
本领域技术人员可以借鉴本文内容,适当替换和/或改动工艺参数实现,然而特别需要指出的是,所有类似的替换和/或改动对本领域技术人员来说是显而易见的,它们都被视为包括在本发明。本发明所述内容已经通过较佳实例进行了描述,相关人员明显能在不脱离本发明内容、精神和范围内对本文所述的内容进行改动或适当变更与组合,来实现和应用本发明技术。
[0064]
应该指出,以下详细说明都是示例性的,旨在对本技术提供进一步的说明。除非另有指明,本文使用的所有技术和科学术语具有与本技术所属技术领域的普通技术人员通常理解的相同含义。
[0065]
需要注意的是,这里所使用的术语仅是为了描述具体实施方式,而非意图限制本技术的技术方案。如在这里所使用的,除非上下文另外明确指出,否则单数形式也意图包括复数形式,此外,还应当理解的是,当在本说明书中使用术语“包含”和/或“包括”时,其指明存在特征、步骤、操作、器件、组件和/或它们的组合。
[0066]
本发明中,舌苔微生物信息具体包括微生物属、种及其丰度,凡是能够获得试样舌苔微生物属、种及其丰度的方法均可用来获得相关信息,本技术采取下述方法获得微生物信息:
[0067]
试样(受试者,包括训练集合测试集)食用早餐和水之前,以无菌水漱口3次,专业操作人员使用舌苔拭子收集其舌苔样本,具体是用咽拭子同时滚动拭子,从舌根部到尖端刮舌30次(每个拭子滚动5次,共6个拭子),取样完成后立即将拭子放入冷冻管中,然后将样品转移到-80℃的冰箱中;
[0068]
按照制造商的说明,应用e.z.n.a的组织dna提取试剂盒(d3396-01;omega,norcross,georgia,usa)提取微生物dna。应用axyprep的pcr纯化试剂盒(ap-pcr-500g;corning,ny,usa)分离、提取和纯化pcr产物,并使用quant-it picogreen dsdna试剂(p7581,thermo scientific,waltham,ma,usa)对产物进行定量测定,最后通过lc-bio co.,ltd的novaseq测序仪进行2
×
250bp末端测序。
[0069]
以下详细描述本发明。
[0070]
《临床标本》
[0071]
进行了全国性多中心临床研究消除了地域、饮食、中心差异对研究的影响,包括8个城市的11个中心,分别位于东部的杭州、温州和上海,南部的福州,西部的成都,北部的辽
宁和黑龙江,中部地区的太原。
[0072]
如图1所示,从2020年1月至2021年10月,从8个中心招募了1111名胃癌(gc)患者,从3个中心招募了1519名非胃癌(ngc)患者,包括169名健康对照(hcs)、648名浅表性胃炎(sgs)和702名萎缩性胃炎(ags)。胃癌(gc)患者中选取865例、非胃癌(ngc)患者中随机选取1287例对前述系统进行训练和验证,其中早期gc(tnmi ii期)448例,晚期gc(tnmiii iv期)417例)、健康对照组(hc)141例、浅表性胃炎(sg)547例、萎缩性胃炎(ag)599例;大约80%的案例被用作训练数据集,大约20%的案例被用作内部验证数据集。此外,将来自3个中心的246例gc和232例ngc作为独立的外部验证数据集,包括162例早期gc、84例晚期gc、28例hc、101例sg和103例ag。这些胃癌(gc)患者均为新诊断胃癌,既往未接受过针对其疾病的治疗,也未针对其疾病进行手术、化疗、放疗、靶向治疗或生物治疗。胃癌(gc)患者均未单发肿瘤,即发现患有两种或多种恶性肿瘤的患者也被排除在外。hcs、sgs和ags经胃镜检查证实。
[0073]
收集所有参与者的临床信息,包括年龄、性别、身高、体重、家族史、吸烟、饮酒、tnm分期、血液肿瘤标志物等。病理分期基于美国癌症联合委员会第8版第23期。表1中示出了gc组和ngc组之间的一般患者信息,例如年龄、性别、bmi、吸烟和饮酒情况,无论是在训练、内部验证还是独立的外部验证数据集中都非常匹配。
[0074]
表1、gc和ngc参与者的临床信息
[0075][0076]
《统计分析》
[0077]
所有统计分析均使用spss23.0软件(spssinc.,chicago,il,usa)进行。结果表示为平均值
±
sd或平均值
±
sem。根据数据是否呈正交分布,使用参数检验或非参数检验。计数数据采用卡方检验分析。p《0.05被认为具有统计学意义。
[0078]
《伦理批准》
[0079]
本技术研究获得了11家参与中心(irb-2019-56)使用的集中伦理委员会的批准,
具体包括浙江省肿瘤医院研究伦理委员会、温州医科大学第一附属医院、辽宁省肿瘤医院、上海交通大学附属仁济医院、福建省肿瘤医院、哈尔滨医科大学附属肿瘤医院、四川省肿瘤医院、山西省肿瘤医院、浙江省同德医院、浙江省中医院、余杭市人民医院。
[0080]
《临床验证》
[0081]
实施例1:
[0082]
利用计算机辅助手段,设计了一种基于深度学习的肿瘤预测系统,该系统根据不同属、种下的微生物丰度自动化预测不同测试试样属于肿瘤阳性的概率。
[0083]
我们分别从胃癌患者和非胃癌人群中获得阳性和阴性试样的舌苔微生物属、种丰度数据。
[0084]
基于上述舌苔微生物属、种丰度数据,设计了一种基于多层感知器(mlp)的神经网络,通过将舌苔微生物在不同属、种上的丰度作为输入,最终依据所提取的高维特征来预测测试试样属于阳性的概率。
[0085]
整体算法结构如图2所示,整个模型的输入是长度为706或1339的向量,分别代表微生物属或种的数量,输入向量的每一个元素代表在特定属或种上的微生物丰度。多层感知器的输入层和输入向量长度对应。通过添加隐藏层提取微生物丰度的高维特征。输出层包含了两个神经元和softmax分类器,分别输出输入样例为胃癌阳性和阴性的概率,各层神经元之间分别互相连接,利用输出的概率分布最小化交叉熵目标函数:
[0086][0087]
其中yi是测试试样对应的真实one-hot标注向量中的元素,是模型预测为类别yi的概率,其中0代表阴性类别,1代表阳性类别。one-hot标注是0、1向量形式的标注,比如分两个类别,类别0,1所对应的one-hot形式的标注是(1,0,0),(0,1,0)。
[0088]
从浙江省肿瘤医院和浙江中医药大学第一附属医院收集328个gc(胃癌)和304个ngc(非胃癌,包括155个hcs(健康)和149个ags(萎缩性胃炎))的舌苔微生物信息数据,胃癌参与者与非胃癌参与者的临床信息如表2所示,可知两组的年龄、性别、吸烟和饮酒等一般临床信息匹配良好。
[0089]
表2、舌苔参与者的临床信息
[0090]
临床指标胃癌(n=328)非胃癌(n=304)p值年龄(岁)63.747
±
11.2563.82
±
6.220.923性别(女/男)114/214123/1810.139吸烟(是/否)223/105218/860.309饮酒(是/否)219/109223/810.071
[0091]
胃癌参与者与非胃癌参与者按照8:2的比例将数据划分成训练集和测试集。在测试集上的敏感性、特异性和准确性如表3和表4所示。
[0092]
表3、测试结果(属)
[0093]
微生物组敏感性特异性准确率属0.9140.9470.929
[0094]
表4、测试结果(种)
[0095]
微生物组敏感性特异性准确率种0.9290.9470.937
[0096]
由表3和表4可知,对于胃癌的诊断预测,舌苔微生物组在属和种两个层级均表现相同的高特异性,其数据相同的可能原因是样本量有限,应当看到微生物组在种层级的敏感性和准确率均高于属层级,均表现出对于胃癌诊断预测的高敏感性、特异性和准确率,作为对比,将基于舌苔微生物的肿瘤检测系统与常规临床应用的血液肿瘤标志物进行比较,选用多种经典血液肿瘤标志物的组合验证对于肿瘤的预测,可供选择的血液肿瘤标志物选自甲胎蛋白(afp)、癌胚抗原(cea)、癌抗原125(ca125)、癌抗原15-3(ca15-3)、癌抗原199(ca199)、癌抗原72-4(ca72-4)、癌抗原242(ca242)、癌抗原50(ca50)、非小细胞肺癌相关抗原(cyfra21-1)、小细胞肺癌相关抗原(神经元特异性烯醇化酶,nse)、鳞状细胞癌抗原(scc)、总前列腺特异性抗原(tpsa)、游离前列腺特异性抗原(fpsa)、α-l-岩藻糖苷酶(afu)、eb病毒抗体(ebv-vca)、肿瘤相关物质(tsgf)、铁蛋白(ferritin)、β2-微球蛋(β2-mg)、胰胚胎抗原(poa)或胃泌素前体释放肽(progrp)中的至少一种,特别是选自cea、ca242、ca72-4、ca125、ca199、ca50、afp或ferritin中的至少一种,更特别是选用上述八种血液肿瘤标志物的组合。基于上述所述血液肿瘤标志物指标的预测方法包括下述步骤:
[0097]
1)数据预处理:由于所有病例的血清指标存在不同程度的缺失,而训练数据需要完整。因此在模型训练之前首先需要对数据进行补全,本技术采用k近邻缺失值插补法对数据进行补全;具体地,缺失的血清指标补全值为2个最近邻居的值的平均值;
[0098]
2)模型训练:本发明采用三种机器学习分类方法,其分别为支持向量机(svm),决策树(dt)和k-近邻分类器(knn),具体地,病例的八种血液肿瘤标志物指标(cea、ca242、ca72-4、ca125、ca199、ca50、afp和ferritin)对应样本特征,病例的阴阳性诊断对应样本的标签,所有补全后的样本均送入三种分类器进行拟合;
[0099]
3)模型评估:本技术采用了内部验证和外部验证对模型进行了评估;内部验证采用与训练数据相同医院、不同病例的数据,而外部验证采用了与训练数据不同的医院病例数据。采用包括敏感性,特异性和准确性在内的三种指标对模型进行预测。
[0100]
相关gc病患的血液肿瘤标志物临床信息如表5所示,可知与ngc患者相比,gc患者的cea、ca424、ca724、ca125、ca199、ca50、afp和ferritin等血液肿瘤标志物浓度显著升高。
[0101]
表5、gc病患的血液肿瘤标志物临床信息
[0102][0103]
大约80%的案例被用作训练数据集,大约20%的案例被用作内部验证数据集,将来自3个中心的246例gc和232例ngc作为独立的外部验证数据集。基于血液肿瘤标志物的机器学习分类方法对于gc诊断的敏感性、特异性、准确性验证结果见表6所示,基于血液肿瘤标志物的三种模型svm、dt、knn的内部验证敏感性介于0.283-0.566,特异性介于0.688-0.976,准确性介于0.603-0.622,而外部验证敏感性介于0.362-0.539,特异性介于0.759-0.938,准确性介于0.645-0.662;基于血液肿瘤标志物的机器学习分类方法对gc诊断在内部验证和外部验证的roc及auc参见图3,可知内部验证的auc值范围为0.682至0.715,外部验证的auc值范围为0.694至0.760;可知在svm算法中,内部验证和外部验证的特异性均达到90%以上,说明该算法能够为胃癌诊断提供有价值的信息。而在dt和knn中,特异性有所下降,敏感性和准确性均有不同程度的提升,可以为胃癌诊断提供多方面的信息。
[0104]
表6、基于血液肿瘤标志物的模型对gc诊断的敏感性、特异性、准确性
[0105][0106]
应当明确的是,本技术的上述对比方案选用了cea、ca242、ca72-4、ca125、ca199、ca50、afp和ferritin在内的八种血清指标,增加、缩减或者代替若干种血清指标均可对肿瘤特别是胃癌的阴阳性进行预测。上述对比方案采用了三种机器学习分类器svm、dt和knn,采用其他机器学习分类器方法如逻辑回归、随机森林亦可达到相应的目的。
[0107]
对比分析表3-4与表6可以看出,可知本技术提供的基于舌苔微生物的肿瘤预测系统具有显著优于上述基于血液肿瘤标志物模型的敏感性和准确率,而且特异性优于模型dt和knn,本技术方案前瞻性强、经济性好、无创伤、敏感性、特异性和准确性更高。
[0108]
主坐标分析(principal coordinate analysis,pcoa)是一种将多维数据进行降
维以研究数据相似性或差异性的可视化方法,基于距离矩阵来寻找主坐标,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,有效地找出数据中最“主要”的元素和结构,对样本之间的关系进行描述,首先随机取样计算各样本之间的单面距离,再根据距离矩阵绘制二维pcoa图,因此若样本的距离越接近,即微生物的丰度和构成越相似,那么它们在pcoa图中的距离越近,gcs组和ngcs组的微生物的主坐标分析图如图4所示,gcs组和ngcs组前30个微生物的热图如图5所示,分析图4和图5可知,gcs组及ngcs组的微生物组之间存在着显著差异。
[0109]
香农多样性指数是用于调查群落局域生境内多样性(α-多样性)的指数,其值与物种多样性呈现正相关,本技术香农多样性指数如图6所示,表明在α多样性方面,gcs的物种丰富度显着高于ngcs,表明gcs舌苔中的物种数量较多。
[0110]
此外,我们使用80%的样本作为训练集,20%的样本作为验证集,建立mlp模型,图7示出了验证集的roc(receiver operating characteristic)和auc(area under roc curve),可知本技术系统具有较为远离(0,0)-(1,1)连线的roc曲线,并观察得知auc属的值为0.945,种的值为0.975,种/属的auc值均显著的高于八种血液肿瘤标志物的svm、dt和knn模型的内部验证auc值(0.682-0.715)和外部验证auc值(0.694-0.760),可知本技术基于舌苔微生物的肿瘤预测系统是一个表现较好的预测模型,对gc的诊断价值明显优于单纯应用八项血液肿瘤标志物的组合的模型。
[0111]
对比gcs组和ngcs组的物种分布,在属水平的显示如图8的e所示,在种水平的显示如图8的f所示,可知本技术基于舌苔微生物的肿瘤预测系统可依据舌苔微生物在属和种的层面上清晰地区分从而对gcs进行针对性的诊断预测,提供了一种针对于肿瘤的前瞻性的、经济性的、无创性的、有效性的筛查和诊断预测方法。
[0112]
上述实施例中的常规技术为本领域技术人员所知晓的现有技术,故在此不再详细赘述。
[0113]
本文中所描述的具体实施例仅仅是对本发明精神作举例说明。本发明所属技术领域的技术人员可以对所描述的具体实施例做各种修改或补充或采用类似的方式替代,但并不会偏离本发明的精神或者超越所附权利要求书所定义的范围。
[0114]
尽管对本发明已作出了详细的说明并引证了一些具体实施例,但是对本领域熟练技术人员来说,只要不离开本发明的精神和范围可作各种变化或修正是显然的。
[0115]
以上所述仅为本技术的优选实施例而已,并不用于限制本技术,对于本领域的技术人员来说,本技术可以有各种更改和变化。凡在本技术的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本技术的保护范围之内。
[0116]
本发明未尽事宜均为公知技术。
当前第1页1
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!